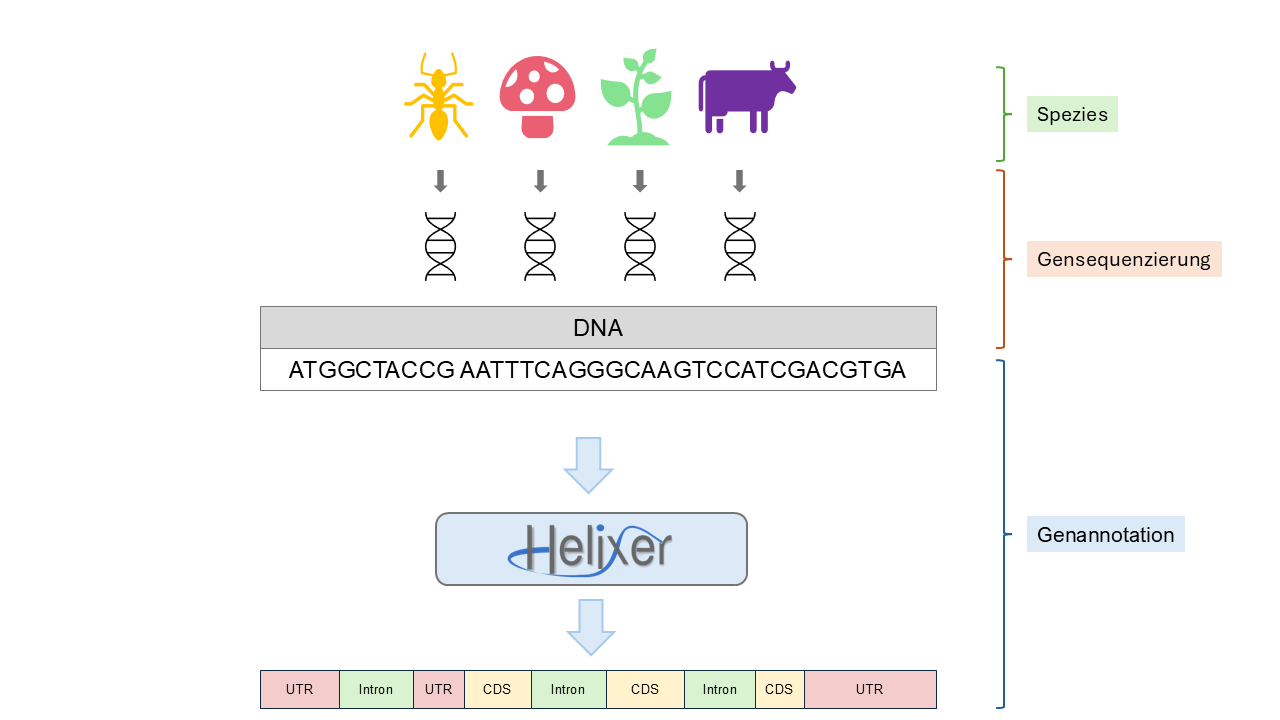
Bevor Biologinnen und Biologen Aussagen über die genetischen Eigenschaften eines Organismus treffen können, müssen sie zunächst wissen, wo sich die Gene in der langen Abfolge der DNA-Bausteine befinden. Dieser Prozess, die sogenannte strukturelle Genannotation, gehört zu den anspruchsvollsten Schritten der Genomanalyse. Bislang waren dafür umfangreiche experimentelle Daten oder gut untersuchte verwandte Arten notwendig. Das KI-Tool Helixer vereinfacht und beschleunigt diesen Schritt nun erheblich.
Die KI erkennt typische Merkmale von Genen direkt aus den Genomsequenzen. Dazu gehören etwa Start- und Stoppsignale – sie steuern die die Auslese einzelner Gene –, die sogenannten nicht-translatierten Bereiche („untranslated region“, kurz UTR), die nicht in Proteine übersetzt werden sowie strukturelle Elemente wie kodierende DNA-Sequenzen („coding DNA sequence“, kurz CDS) – sie enthalten die bekannten Bauanleitungen für Proteine – und schließlich die „Introns“ genannte Zwischenstücke.
„Es ist, als würde man in einem völlig unbekannten Buch plötzlich Absätze, Kapitel und einzelne Wörter erkennen“, erklärt die Korrespondenzautorin der in Nature Methods veröffentlichen Studie Marie Bolger vom Jülicher Institut für Bioinformatik (IBG-4; Direktor: Prof. Dr. Björn Usadel). „Das macht die Genomforschung deutlich schneller.“
Ähnlich präzise wie klassische Verfahren
Helixer basiert auf einem sogenannten Deep-Learning-Ansatz, einer Methode des maschinellen Lernens, die auf künstlichen neuronalen Netzen basiert. Es ist das Produkt enger Zusammenarbeit zwischen drei Arbeitsgruppen an der HHU und dem Forschungszentrum Jülich. Dr. Alisandra Denton entwickelte am Institut für Biochemie der Pflanzen der HHU zunächst gemeinsam mit Düsseldorfer Studierenden den Deep-Learning-Ansatz von Helixer. Prof. Dr. Björn Usadel und sein Team erweiterten Helixer dann um ein Hidden Markow Modell, welches die basengenaue Definition von genomischen Features erlaubt. Auch Prof. Dr. Oliver Ebenhöhs Arbeitsgruppe vom Institut für Quantitative und Theoretische Biologie der HHU war an der Programmierung von Helixer beteiligt.
Dr. Alisandra Denton, die heute in Kanada arbeitet, erinnert sich: „Während meiner Postdoc-Zeit im Labor von Prof. Dr. Andreas Weber an der HHU hatte ich viel Freiheit, Dinge zu erforschen, zu lernen und auszuprobieren. Damals waren wir in der Anfangsphase des KI-Einsatzes für die Genomanalyse. Helixer war zunächst ein exploratives Projekt, eine echte Übung im ‚Learning by Doing‘. Es ist spannend zu sehen, wie nützlich Helixer heute ist und wie häufig es jetzt genutzt wird. Es hat den Fortschritt in diesem Bereich beschleunigt.“
Heute ist Helixer ein Tool, das praktisch nutzbare Ergebnisse erzielt. Es ist das erste KI-Werkzeug, das Gene in sehr unterschiedlichen Organismengruppen – von Pflanzen und Pilzen über Insekten bis hin zu Wirbeltieren – zuverlässig bestimmen kann. Weltweit werden jedes Jahr Tausende Genome entschlüsselt, viele davon von bislang kaum erforschten Arten. Für sie kann Helixer nun sofort nutzbare Geninformationen liefern, für die zuvor oft monatelange Analysen erforderlich waren.
Die KI sagt die Grenzen von Genen voraus und erreicht dabei nahezu die Qualität manuell kuratierter Referenzannotationen – ganz ohne zusätzliche Daten. Bei Wirbeltieren erzielt Helixer eine hohe Genauigkeit und übertrifft etablierte Genprädiktionswerkzeuge über ein breites Artenspektrum hinweg. Insbesondere bei Pflanzen zeigt Helixer eine deutlich bessere Vorhersage der Genstruktur.
Neue Impulse für das Forschungsfeld
„Wir konnten zeigen, dass Helixer für eine Vielzahl von Organismen funktioniert, was für den Einsatz in der Pflanzenzüchtung, Biotechnologie und Umweltforschung entscheidend ist“, betont Bolger. „Diese Fortschritte in der KI-gestützten Genannotation sind für das Fachgebiet wirklich spannend.“
Die Genomsequenzierung wurde bereits vor mehr als 20 Jahren automatisiert und hat seither eine enorme Datenfülle hervorgebracht. Die Genannotation galt dagegen lange Zeit als Flaschenhals der Genomanalyse. „Fast zwei Jahrzehnte lang gab es in diesem Bereich keine grundlegend neuen Ansätze“, sagt Prof. Dr. Björn Usadel vom FZJ, der auch Professor an der HHU ist. „Helixer zeigt, dass moderne KI-Methoden helfen können, diesen Engpass zu überwinden.“
In der Fachgemeinschaft hat Helixer viel Resonanz erfahren. „Wir sehen, dass das Tool in vielen Projekten eingesetzt wird – von Nutzpflanzen bis hin zu Insekten, die ganze Ökosysteme prägen“, sagt Usadel.
Originalpublikation
Felix Holst, Anthony M. Bolger, Felicitas Kindel, Christopher Günther, Janina Maß, Sebastian Triesch, Niklas Kiel, Nima Saadat, Oliver Ebenhöh, Björn Usadel, Rainer Schwacke, Andreas P. M. Weber, Marie E. Bolger & Alisandra K. Denton; Helixer: ab initio prediction of primary eukaryotic gene models combining deep learning and a hidden Markov model; Nature Methods (2025)